|
L&M-6-2012
>
Molekulargenetische Diagnostik von erblichen Netzhauterkrankungen
Molekulargenetische Diagnostik von erblichen NetzhauterkrankungenSchau mir in die Augen, Kleines …Mit seinem berühmten Satz in „Casablanca“ ging es Humphrey Bogart nicht um Netzhautdiagnostik. Anders jedoch Wissenschaftlern der Universität Regensburg: Mit einem tiefen Blick in die genetische Welt erkennen sie frühzeitig mit Mikrochip-Elektrophorese und neuer RetChip-Technologie erbliche Netzhauterkrankungen. Sehen ist neben Hören, Riechen, Schmecken und Tasten eine der fünf Sinnesleistungen, die für die meisten Menschen selbstverständlich zum Alltag dazugehören. Wenn das Sehen jedoch plötzlich nur noch eingeschränkt möglich ist, werden viele Kleinigkeiten des Alltags für den Betroffenen zu einer großen Herausforderung. Um die Diagnostik und Behandlung dieser Patienten zu vereinfachen, wurde am Institut für Humangenetik der Universität Regensburg ein neues Verfahren entwickelt, das zu neuen Erkenntnissen in Forschung und Diagnostik genutzt wird. Erbliche Netzhauterkrankungen
Es gibt viele unterschiedliche Gründe, warum es zu einer Sehstörung kommen kann und es gibt ebenso zahlreiche unterschiedliche Krankheitsbilder. Umweltbedingungen wie z.B. Rauchen und Übergewicht erhöhen das Risiko, eine häufige Netzhautkrankheit wie die altersabhängige Makuladegeneration zu bekommen, jedoch betonen neueste Forschungsergebnisse vor allem auch den genetischen Ursprung solcher Krankheiten. Der RetChip v1.0 – gleichzeitiger Test verschiedener Gene
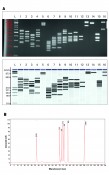
Um die genetische Ursache einer Netzhauterkrankung nachzuweisen, hat das Institut für Humangenetik Regensburg den so genannten RetChip v1.0 entwickelt. Hierbei können bis zu 72 potenziell defekte Gene parallel analysiert werden. Die klinische Fragestellung legt die zu testenden Gen-Sets („Module“) vor. So können z.B. in dem „Modul Morbus Stargardt“ die Gene, die für die Stargardt-Erkrankung verantwortlich sind, getestet werden, wie z.B. das Gen ABCA4.Die neue RetChip-Technologie erlaubt es – im Vergleich zu konventioneller klinischer Diagnostik – viele Gene gleichzeitig zu testen und steigert so die Wahrscheinlichkeit, die Ursache der Erkrankung zu identifizieren. Zudem können nun auch neue, noch nicht bekannte Veränderungen in diesen Krankheitsgenen identifiziert werden. Eine durch den RetChip gefundene Veränderung der DNA-Sequenz wird mittels Sanger-Sequenzierung bestätigt. Die so genannten Soll-DNA-Sequenzen „Standard“-DNA-Sequenzen ohne Gendefekt) befinden sich auf dem Chip. Die gefärbte Patienten- Automatisierte Multiplex-Analysen Um die richtigen Mengen an DNA zu bestimmen, die für die Analyse des RetChip v1.0 eingesetzt werden müssen, wurden diese Multiplexe früher durch Gel-Elektrophorese untersucht (Abb. 1). Da dies jedoch viel Zeit und manuelle Arbeit erforderte, wird diese Methode zukünftig immer öfter durch automatische Elektrophoresesysteme ersetzt. Das MCE-202 MultiNA-Gerät von Shimadzu (Abb. 2) bietet bei der Analyse der DNA-Fragmente eine moderne Alternative zur Agarose-Gelelektrophorese. Manuelle Arbeitsschritte für Vorbereitung, Trennung, Nachweis und Datenaufbereitung laufen komplett automatisiert ab. Die Größenbestimmung und die Quantifizierung von DNA-Fragmenten basieren auf einer Mikrochiptechnologie, wobei wiederverwendbare Quarzmikrochips eingesetzt werden. Fazit Durch den Nachweis der genetischen Ursache mithilfe der Multiplex-PCR, Mikrochiptechnologie und RetChip-Analyse kann die klinische Diagnostik verlässlich durchgeführt werden. Der Patient kann nun besser auf den zukünftigen Krankheitsverlauf vorbereitet und in einer gezielten Therapie ggf. medikamentös oder mit bekannten Nahrungsergänzungsmitteln behandelt werden. Betroffene Familien können dahingehend beraten werden, wie hoch das Risiko ist, dass andere Familienangehörige und deren Nachkommen an dieser Krankheit erkranken (Wiederholungsrisiko). Zudem wird die Forschung auf diesem Gebiet vereinfacht. Die zunehmenden Erkenntnisse in der Molekulargenetik steigern damit auch die Wahrscheinlichkeit für eine künftige sinnvolle Gentherapie.
Abb.1: Abb.2: MCE-202 MultiNA-Gerät Quellen:
Mellough C.B., Steel D.H.W. und Lako M. (2009): Genetic Basis of Inherited Macular Dystrophies and Implications for Stem Cell Therapy Stem Cells, 27(11): 2833–2845. Foto: © Vanessa Liedschulte |
L&M 6 / 2012
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Der Autor:Weitere Artikel online lesenNewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |