|
L&M-3-2008
>
PCR - Molekulare Sepsisdiagnostik
PCR - Molekulare SepsisdiagnostikAbreicherung humaner DNA aus Blut erhöht die Sensitivität der PCR-Detektion von Sepsiserregern
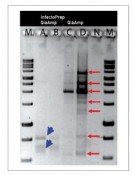
Dr. Claudia Disqué, Hochschule Bremerhaven Eine viel versprechende Methode für den generellen Nachweis von Erregern ist die qPCR-Reaktion unter Verwendung von Primern für hoch konservierte Regionen in 16Soder 23S rRNA- Genen. Die Amplifikate können zur Differenzierung auf Nukleinsäureebene herangezogen werden. Durch Sondenhybridisierungen an variable Regionen im Gen kann Auskunft über die Gram-Zugehörigkeit, Familie, Gattung bis hin zur Spezies erhalten werden [1, 2].Die Sequenzierung des Amplifikats und Online-Datenbankrecherche(z.B. BLAST, FASTA) ist eine andere, direkte Methode zur Identifizierung eines Erregers. Wichtig für das Design von Primern ist, Spezifität für das zu amplifizierende Target zu erreichen. Bei generellen16S rRNA-Gen-gerichteten Primern ist es erforderlich, Regionen zu finden, die alle Bakterien erfassen (pan-bakteriell)und gleichzeitig gegen andere Organismengruppen(z.B. Hefen und andere pathogene Pilze) diskriminieren. So konstruierte Primer werden dann anhand einer möglichst großen Auswahl von Bakterien validiert [3]. Weniger Beachtung in der Diagnostik von Sepsiserregern findet bisher die Möglichkeit, dass Primer unspezifische Reaktionen auch mit der humanen DNA eingehen können [4].Als Konsequenz kann es zu einem Verlust an Detektionssensitivität und -spezifität des Assays kommen. Störfaktor humane DNA Die Erregerzahlen in Blut von Sepsispatienten können sehr niedrig sein (1–100 KBE/ml, 5). Umgerechnet auf DNA-Masse (ca. 5 fg/E. coli-Zelle) entspricht das einem2 • 107 bis 2 • 109-fachen Überschuss humaner DNA (typischer Gehalt: ca. 10 µg/ml). Angesichts der Größe des humanen Genoms ist zu erwarten, dass unspezifische Reaktionen zwischen bakterienspezifischen Primern und humanen Sequenzen auftreten und zu falschen Signalen führen können. In Abb. 1 ist eine PCR mit einem universellen16S rDNA-Primerpaar gezeigt. In dem Versuch traten unspezifische Amplifikate bereits bei einem nur 3.200-fachen Überschuss humaner zu bakterieller DNA auf(Abb. 1, PCR, b). Mit weiterer Abnahme der bakteriellen DNA wurde die humane Bande stetig stärker, was bedeutet, dass die Amplifikation des unspezifischen gegenüber des spezifischen Signals immer weiter überwog. Humane DNA alleine resultierte in einer deutlichen Bande (Abb.1, PCR, c), die bei Sequenzierung als ein Abschnitt eines humanen Genes (HNRPA1 Pseudogen) identifiziert werden konnte (Abb. 1, siehe PCR, b). Im Versuch mit Mischungen bakterieller und humaner DNA kam es bei der Sequenzierung zu Bandenüberlagerungen (Abb. 1, Sequenzierung,b), die eine Identifizierung durch BLAST Analyse unmöglich machten. Diese Versuche zeigen anschaulich, dass Kreuzreaktionen der universellen 16SrDNA-Primer mit humanen Sequenzen auftreten können. Als Resultat kann es zu unspezifischer Amplifikationkommen, was zum Verlust an Spezifität und zur falschpositiven Befundung führt. Zudem können Sequenzüberlagerungen die Identifizierung von Erregern in der Sequenzanalyse erschweren bzw. unmöglich machen. Unspezifische Reaktionen der Primer können auch zu einem Verlust an Sensitivität der Detektion von Erregern beitragen. Das geschieht dann, wenn Primer in größerem Abstand zueinander oder in die gleiche Richtung an humane DNA binden und einzelsträngige Amplifikate in einer linearen Reaktion entstehen. Bei solcherart Austitrierung ist die Primermenge für die spezifische Reaktion mit bakteriellen Sequenzen limitiert. In einem Experiment mit Mischungen reiner DNAs aus Bakterien und humanem Blut wird dieser Effekt verdeutlicht(Abb. 2). Auffällig ist, dass die humane DNA diecrossing points (C(t)-Werte) im Mittel um 7,7 Einheiten verschob, also umgerechnet die Detektion des Erregers um mehr als zwei Zehnerpotenzen herabsetzte. Beseitigung der humanen DNA während der DNA-Präparation erhöht die Detektionssensitivität in der qPCR
Ansätze, dem negativen Effekt humaner DNA auf die universelle16S rDNA PCR durch gezielte Konstruktion von Primern entgegen zu wirken, stößt auf Einschränkungen der begrenzten Zahl von konservierten Bereichen im Gen. Tatsächlich konnte hier durch die Verwendung eines anderen, gängigen Primerpaares (siehe 3) die Entstehung unspezifischer Amplifikate nicht verhindert werden. Eine neue Strategie ist, humane DNA während der Extraktion abzureichern und damit nicht erst in die PCR einzubringen. Dieser Ansatz wird mit einem Verfahren(DNA InfectoPrep, AppliChem) verfolgt, nach dem die Blutzellen lysiert, die freigesetzte humane DNA degradiert, die Erregerzellen angereichert und die Bakterien- DNA isoliert wird. Fazit Die neue Methode für die Beseitigung humaner DNA während der DNA-Extraktion ist eine leistungsfähige und vielversprechende Methode, um hohe Sensitivität und Spezifität in der Sepsisdiagnostik mittels universeller 16SrDNA PCR zu erreichen. Das Verfahren muss allerdings inklinischen Studien im Vergleich mit der Blutkultur diagnostik noch validiert werden, um es in der Routine einsetzen zu können. Foto: © applichem.de
[1] Klaschik, S., Lehmann, L.E., Raadts, A., Book, M., Gebel, J., Hoeft, A., Stuber, F. |
L&M 3 / 2008
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Weitere Artikel online lesenNewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |