|
Lichtmikroskopie jenseits des Abbe-Limits
Lichtmikroskopie jenseits des Abbe-LimitsBlinkende MoleküleMit dem diesjährigen Nobelpreis in Chemie ehrt das Nobelpreiskomitee die Erfinder von Superauflösungs-Mikroskopiemethoden. Die Entdeckungen von Eric Betzig, Stefan Hell und William Moerner waren in den letzten Jahren Grundlage für bahnbrechende Forschungsergebnisse in den Lebenswissenschaften, die mit herkömmlichen Fluoreszenz-Mikroskopie- methoden nicht möglich waren.
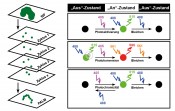
Abb.1 Die Abbe-Formel beschreibt die durch Beugung limitierte erreichbare Auflösung in der klassischen Lichtmikroskopie. Diese Grenze wird als Auflösungsgrenze oder Abbe-Limit bezeichnet.
Die fotoaktivierte Lokalisationsmikroskopie (Photoactivated Localization Microscopy, PALM) wurde 2006 von Eric Betzig, Harald Hess und Kollegen entwickelt, um die Position einzelner Moleküle mit einer Auflösung jenseits der von Ernst Abbe beschriebenen optischen Auflösungsgrenze zu bestimmen (Abb.1). Heute forschen beide am Howard Hughes Medical Institute des Janelia Research Campus in Ashburn, Virginia, USA. Wenn in der Lichtmikroskopie fluoreszierende Proteine wie z.B. GFP zu nahe beieinanderliegen, können feine zelluläre Strukturen nicht mehr getrennt voneinander abgebildet werden, sie erscheinen als „Wolke“. PALM und verwandte Superauflösungs-Techniken (u.a. FPALM, STORM) nutzen daher eine Reihe von Tricks, um die maximal mögliche Auflösung in der Fluoreszenzmikroskopie erheblich zu steigern. Wichtigste Voraussetzung hierfür sind sogenannte fotoschaltbare fluoreszierende Proteine (photoswitchable fluorescent proteins, PS-FPs). In der Molekularbiologie werden diese PS-FPs an zelluläre Strukturen gebunden und selektiv zum „Blinken“ gebracht. Mit geringen Lichtintensitäten und spezifischen Wellenlängen werden nun sequenziell einige wenige PS-FPs zufällig aktiviert, das heißt auf „An“ gesetzt. Im Anschluss wird ein Bild mit einer leistungsfähigen Mikroskopkamera aufgenommen. Dadurch ist die Wahrscheinlichkeit, dass zwei eng benachbarte Moleküle gleichzeitig aufleuchten, sehr klein. Bereits leuchtende Moleküle werden durch Licht geeigneter Wellenlänge wieder inaktiviert, das heißt, sie gehen in den „Aus“-Zustand über. Um alle Moleküle zu erfassen, wird dieser Vorgang sehr häufig und schnell hintereinander wiederholt (Abb.2). Dabei können weit über tausend Einzelaufnahmen entstehen, bis alle PS-FPs endgültig in ihren „Aus“-Zustand konvertiert sind. Ab hier übernimmt dann der Computer: Durch spezielle Algorithmen lässt sich der Mittelpunkt der fluoreszierenden Molekül-„Spots“ genauestens lokalisieren. Damit sind die Positionen der einzelnen Moleküle exakt bestimmbar. Das fertige Bild in „Superauflösung“ besteht somit aus der Summe aller detektierten Einzelmoleküle in allen Einzelaufnahmen – ähnlich einem pointilistischen Kunstwerk. Weitere Entwicklungen der PALM-Methode ermöglichen heute 3D-Imaging mit Auflösungen von etwa 20 Nanometern in lateraler und 60 Nanometern in axialer Raumrichtung. Das ist das Zehnfache der mit klassischer Lichtmikroskopie erreichbaren Auflösung (Abb.3). Das Abbe-Limit besteht immer noch, aber es lässt sich austricksen – dank brillanten Nobelpreisträgern wie Eric Betzig und ELYRA-Mikroskopen von ZEISS.
Abb.2 Das Funktionsprinzip von PALM-Mikroskopie
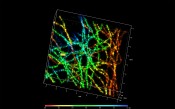
Abb.3 3D-PALM-Mikroskopie von Alpha-Tubulin mit ZEISS ELYRA PS.1 Mit freundlicher Genehmigung von Mike Davidson, Florida State University, USA.
Mehr Informationen // iBiology Seminar mit Eric Betzig & Harald Hess (Englisch) // ZEISS Imaging-System ELYRA // White Paper Sample Preparation for Superresolution Microscopy (Englisch) |
L&M 9 / 2014
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download NewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |