|
Welche genetischen Elemente unsere und andere Genome seit Millionen von Jahren verändern
Welche genetischen Elemente unsere und andere Genome seit Millionen von Jahren verändernMobile Elemente im GenomIn den Genomen vieler Arten leben quasi besondere genetische Elemente, die sich innerhalb des Genoms bewegen oder vervielfältigen können. Seit vielen Millionen Jahren verändern mobile genetische Elemente das sie beherbergende Genom. In manchen Arten machen Reste solcher Elemente den Großteil des Genoms aus. Im Menschen und anderen Arten ist eine Vielzahl klinisch relevanter Mutationen durch die Aktivität mobiler Elemente belegt. Genome sind keineswegs statische Moleküle. Immer wieder kommt es zu kleineren oder größeren, mehr oder weniger gravierenden Veränderungen der Nukleotidabfolge. Einzelne Nukleotide werden verändert – bis hin zu umfangreichen Deletionen oder Umlagerungen von Genombereichen. Wichtig für die Veränderung von Genomen sind mobile genetische Elemente, die quasi in den sie beherbergenden Genomen leben. Mobile genetische Elemente finden sich in praktisch allen Arten, z.B. Bakterien, Pilzen, Wirbellosen, Wirbeltieren, Säugern und im Menschen. Wahrscheinlich seit Milliarden von Jahren gibt es mobile genetische Elemente, die während dieser Zeit massiv auf die Struktur der Genome aller möglicher Arten Einfluss genommen haben. In diesem Artikel soll insbesondere auf die mobilen genetischen Elemente im menschlichen Genom eingegangen werden, die insgesamt ungefähr 45% des menschlichen Genoms einnehmen und oftmals sehr ähnlich in den Genomen vieler anderer Säuger bzw. Wirbeltiere vorkommen. Versionen mobiler genetischer Elemente Mobile genetische Elemente können sich auf verschiedene Weisen in Genomen bewegen. Erstens gibt es Elemente, die sich über eine DNA als Zwischenstufe verschieben. Die Elemente, als DNA-Transposons bezeichnet, codieren mindestens für ein Protein, die Transposase, welches das mobile genetische Element als DNA-Fragment präzise aus dem längeren DNA-Strang des Genoms ausschneiden und an anderer Stelle im Genom wieder einsetzen kann. Im menschlichen Genom gibt es keine sich aktiv bewegenden DNA-Transposons mehr. Die noch als solche – also anhand ihrer Sequenzen – mit bioinformatischen Methoden identifizierbaren DNA-Transposons sind viele Millionen Jahre alt, stammen also aus einer Zeit, in der DNA-Transposons in den Genomen unserer evolutiven Vorfahren „lebten“. Die Sequenzreste dieser DNA-Transposons werden seit dieser Zeit in den Genomen von Generation zu Generation einer Art bzw. von Art zu Art vererbt. Weitere Gruppen von mobilen genetischen Elementen bewegen sich über die Zwischenstufe eines RNA-Moleküls. Diese Elemente werden als Retrotransposons bezeichnet [1]. Ein im Genom befindliches Retrotransposon stellt mit dem Prozess der Transkription ein RNA-Molekül von sich selbst her. Dieses RNA-Molekül wird anschließend an einer recht beliebigen Stelle im Genom in ein DNA-Molekül umgeschrieben und an dieser Stelle in das Genom eingebaut. Das Retrotransposon hat sich also von einer Stelle des Genoms an eine andere Stelle kopiert bzw. amplifiziert. Im menschlichen Genom sowie in einer Vielzahl anderer Genome kann man verschiedene Retrotransposons grundsätzlich anhand ihrer Länge einteilen. Es gibt Gruppen von relativ kurzen und relativ langen Retrotransposons. Die kurzen Retrotransposons, als Short Interspersed Elements (SINE) bezeichnet, sind ungefähr 300 Nukleotide lang. Den häufigsten und wichtigsten Vertreter der SINEs im menschlichen Genom stellen die sogenannten Alu-Elemente dar, von denen sich ungefähr 1Mio. Kopien quer über das Genom verteilt finden und ungefähr 13% davon ausmachen. Alu-Elemente sind vor ungefähr 55 Mio. Jahren in evolutiven Vorläufern der zum Menschen führenden Primatenlinie entstanden, in der Folgezeit haben sich Alu-Elemente massiv im Genom amplifiziert. Es scheint leicht nachvollziehbar, dass solche Amplifikationen von Alu-Elementen im Verlauf der Evolution großen Einfluss auf die Struktur von Genomen gehabt haben müssen. Alu-Elemente benötigen für ihre Aktivität eine andere Gruppe von Retrotransposons, die sogenannten Long Interspersed Elements (LINE), die im Idealfall ungefähr 6.000 Nukleotide lang sind. Die häufigsten (ca. 500.000 Kopien; ca. 17% des menschlichen Genoms), wichtigsten und aktuell in menschlichen Genomen mobilen Vertreter sind die sogenannten L1-Elemente (auch als LINE1 oder KpnI-Elemente bezeichnet). L1-Elemente sind gegenüber Alu evolutiv deutlich weiterentwickelt. Sie können Proteine codieren, die essenziell für die Mobilität der Elemente sind; erstens ein Protein, das L1-RNA binden kann, zweitens ein Protein, das eine sogenannte Reverse Transkriptase ist, die RNA in DNA umschreiben kann sowie eine Endonuklease, die DNA-Stränge recht sequenzspezifisch durchtrennen kann. L1-Elemente codieren also die Enzyme, mit denen sie ihre eigene RNA in DNA umschreiben bzw. an einer Stelle im Genom wieder einsetzen können. Zudem besitzen L1-Elemente einen Promoter-Bereich am 5‘-Ende, an dem sie ihre Transkription starten, sowie einen Bereich am 3‘-Ende, an dem sie ihre Transkription beenden. In den meisten Fällen ist die Umschreibung von L1-RNA in DNA unvollständig und es wird infolge dessen nur ein relativ kurzes Stück des 3‘-Endes eines L1-Elementes als Neubildung eingefügt.
In manchen Fällen retrotransponiert die Proteinmaschinerie von L1 auch andere, Nicht-L1-RNA-Moleküle. So werden neue Kopien der oben beschriebenen Alu-Elemente eigentlich durch L1-Proteine gebildet, wenn sich Alu-RNA und L1-Proteine zusammenlagern. L1-Proteine können auch RNA-Moleküle von regulären zellulären Genen retrotransponieren, also DNA-Kopien von mRNA-Molekülen von Genen bilden. Es entstehen sogenannte prozessierte Pseudogene, von denen im menschlichen Genom bisher mehr als 10.000 identifiziert wurden (http:/ Von Retroviren abstammende Elemente Eine andere Gruppe von mobilen genetischen Elementen drang quasi „von außen“ in Genome ein. Es handelt sich bei diesen Sequenzen ursprünglich um funktionell umfangreiche mobile genetische Elemente, nämlich Retroviren, also Sequenzen, die sich auch zwischen Individuen und verschiedenen Arten bewegen können und im Zielorganismus ihr genetisches Material in das Genom von Zellen einbringen. Im Verlauf der Evolution passierte es immer wieder, dass zu der betreffenden Zeit vorkommende Retroviren ihr genetisches Material in das Genom von Keimzellen als sogenanntes Provirus einbauten. Da das Genom von Keimzellen nach Fortpflanzung letztendlich das Genom des neuen Individuums bildet, wird das neue Individuum in allen Zellen das zuvor in die Elterngeneration eingebaute Provirus tragen und wieder an eine nachfolgende Generation weitergeben können; im Verlauf der Evolution unter Umständen auch an neue Arten. Im Verlauf der Evolution der menschlichen Linie haben ungefähr 35 verschiedene Retroviren ihren Weg in die Keimbahn gefunden und detektierbare Sequenzspuren hinterlassen, die in den meisten Fällen 30, 50 oder noch mehr Mio. Jahre zurückreichen. Da diese von Retroviren abstammenden Sequenzen stabil vererbbare Bestandteile des Genoms sind, werden sie im Fall des Menschen als humane endogene Retroviren (HERV) bezeichnet [2]. HERVs haben ihre Kopienzahlen im Genom für gewöhnlich vergrößert, indem sie z.B. im Keimbahngenom neue Proviren bildeten, ohne den Organismus neu infizieren zu müssen. Ungefähr 8% (ca. 700.000 Loci) des menschlichen Genoms gehen auf die Aktivität von Retroviren zurück. HERV-Proviren sind im Vergleich zu Alu und L1 nochmals komplexer aufgebaut. Im intakten Fall codieren sie Proteine mit RNA-bindenden Eigenschaften, die retrovirale Partikel ausbilden können, weiterhin eine Protease, Endonuklease, reverse Transkriptase, RNaseH sowie ein Envelope-Protein, mit dem Retroviren normalerweise in neue Zellen eindringen. Zusätzlich besitzen Proviren an ihren Enden sogenannte Long Terminal Repeats (LTRs), die regulatorische Einheiten zur Transkription sowie einen Promoter und einen Terminator enthalten. Transkripte eines Provirus werden durch eigene Spleißsignale weiter prozessiert. In den meisten Fällen können HERVs aufgrund des evolutiven Alters von mehreren Millionen Jahren und damit einhergehenden vielfachen Mutationen aber nicht mehr die ursprünglichen retroviralen Proteine codieren (s.Abb.). Konsequenzen der Mobilität Wie oben angedeutet, blieb die Aktivität dieser mobilen genetischen Elemente nicht ohne Folgen für die Struktur und Funktionalität der betreffenden Genome. Auch im Menschen lassen sich aktuell Auswirkungen dieser Aktivitäten feststellen. Die Amplifikation von Alu-Elementen auf insgesamt 1 Mio. führte zur Veränderung von Genom- bzw. Genbereichen, wenn ein Alu-Element sich z.?B. innerhalb eines Genes oder etwa in regulatorischen Bereichen eines Gens neu bildete. Solche Neubildungen waren im einfachsten Fall evolutiv neutral, vielleicht brachten sie in dem einen oder anderen Fall aber einen evolutiven Vorteil für den Organismus mit sich. Das Gleiche gilt für Neubildungen von L1-Elementen – auch wenn oftmals nur ein kurzes Stück des 3‘-Endes eines L1-Elementes neu gebildet wurde (s. Abb.). Im Fall der HERVs konnten Konsequenzen für Gene in mehreren Beispielen dokumentiert werden. Zum Beispiel kann der in einem HERV-LTR gelegene Promoter, wenn er im Genom in der Nähe eines benachbarten zellulären Gens lokalisiert ist, zusätzlich zum geneigenen Promoter alternative Transkripte des Gens beisteuern, die eventuell nur in bestimmten Gewebetypen gebildet werden. Weiterhin sind Fälle gezeigt, in denen eine HERV-Sequenz alternative Transkripte erzeugt, indem sie innerhalb eines Gens lokalisiert ist und manchmal die Transkription des Gens vorzeitig terminiert (polyadenyliert) oder eigene Spleißsignale der HERV-Sequenz das Spleißen des Gentranskriptes verändern. Die während der Evolution erfolgten Veränderungen von Genen waren entweder evolutiv neutral oder in einer bestimmten, meist nicht leicht nachvollziehbaren Weise für den Organismus vorteilhaft.
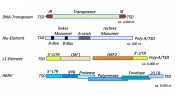
Abb. Aufbau typischer mobiler genetischer Elemente. DNA-Transposons codieren eine Transposase und besitzen am 5‘- und 3‘-Ende ihrer Sequenz sogenannte „invertierte Repeats“ (IR). Das sind gegenläufig-identische Sequenzen, welche die Transposase erkennt und schneidet. Alu-Elemente können in zwei Teilabschnitte (linkes und rechtes Monomer) eingeteilt werden, die von einem Abschnitt reich an A-Nukleotiden getrennt sind. A- und B-Boxen stellen Promoterbereiche dar. L1-Elemente besitzen zwei offene Leserahmen (ORF1, ORF2). die von einer 5‘ und einer 3‘ untranslatierten Region (UTR) flankiert werden. Humane endogene Retroviren haben typischerweise (ehemals) codierende Bereiche für die Gene gag, Protease, Polymerase und Envelope, die von Long Terminal Repeats (LTR) flankiert werden. Zu beachten ist hierbei, dass sowohl L1- als auch HERV-Elemente oftmals nicht in der gezeigten intakten Form vorkommen, sondern mehr oder weniger stark ausgeprägte Deletionen aufweisen. Alu- und L1-Elemente besitzen am 3‘-Ende eine mehr oder weniger lange Abfolge von A-Nukleotiden (Poly-A). Alle gezeigten Elemente sind von sogenannten Target Site Duplications (TSD) flankiert, welche während der Bildung der Elemente entstandene, kurze Wiederholungen der Sequenz der Insertionsstelle sind. Die ungefähren Längen der verschiedenen Elemente sind jeweils in Nukleotiden (nt) angegeben.
Für den menschlichen Organismus sind einige Auswirkungen von mobilen genetischen Elementen funktionell dokumentiert. Zum einen betrifft dies die Aktivität von Alu- und L1-Elementen, die auch aktuell im menschlichen Organismus aktiv sind. Es kommt in vielen Individuen zu Neubildungen von Alu- und L1-Elementen. Projekte zur Sequenzierung von menschlichen Genomen brachten eine Vielzahl von neu gebildeten, sogenannten polymorphen Alu- und L1-Elementen an den Tag, die sich nur in wenigen Verwandten oder vielleicht nur in der betreffenden Person gleichermaßen finden [3]. Manchmal zerstören Neubildungen von Alu- und L1-Elementen Gene mit einem bekannten Phänotyp. So sind z.B. Fälle dokumentiert, in denen die Funktion der (auf dem X-Chromosom lokalisierten) Gene „Faktor VIII“ oder „Faktor IX“ nicht durch die im Kontext von Hämophilien bekannten Mutationen defekt waren, sondern indem sich in einem Individuum ein Alu- oder L1-Element innerhalb dieser Gene während der Embryonalentwicklung neu gebildet hatte, so die Genfunktion zerstört wurde und es deshalb in der Person zur Hämophilie kam. In gleicher Weise sind Fälle von z.B. Apert-Syndrom, Retinitis pigmentosa, zystische Fibrose, Muskeldystrophie Duchenne oder Neurofibromatose als durch die Neubildung von Alu- oder L1-Elementen beschrieben [1]. Wenngleich es schon erste Hinweise gibt, ist die Aktivität von Alu- und L1-Elementen im Kontext der Entstehung von Krebs aktuell Gegenstand der Forschung, indem z.B. in Tumorzellen immer wieder Neubildungen solcher Elemente gefunden wurden, manchmal auch in der Nähe oder innerhalb von tumorrelevanten Genen. Die genaue Relevanz solcher Neubildungen ist weiter aufzuklären [4]. Genauso ist Gegenstand der Forschung, inwieweit von bestimmten HERVs kodierte Proteine an der Entstehung von Tumorerkrankungen, etwa Hodentumoren, beteiligt sein könnten, indem diese HERV-Proteine bekanntermaßen mit funktionell wichtigen zellulären Proteinen interagieren [5]. Relevant statt Junk Mobile genetische Elemente können durchaus positive Konsequenzen für einen Organismus mit sich bringen. Im Menschen gibt es ein von einem HERV-Provirus abstammendes Gen, Syncytin-1, dessen Protein scheinbar essenziell an der Entwicklung der Plazenta beteiligt ist, indem es zu bestimmten Zeitpunkten die Fusion der Zellmembranen bestimmter plazentaler Zelltypen bewirkt. Im Verlauf der Evolution wurde von diesem HERV-Provirus eine ganz spezifische Funktion retroviraler Envelope-Proteine selektiert, die per se eine membranfusionierende Funktion haben. Interessanterweise gab es in verschiedenen Säugetieren ähnliche Entwicklungen, indem auch in anderen evolutiven Linien ein von einem endogenen Retrovirus stammendes Envelope-Protein für die Entwicklung der Plazenta adaptiert wurde – Membranfusionen bestimmter Zellen waren scheinbar mit großen evolutiven Vorteilen verbunden [6]. Vor Jahrzehnten wurden die Sequenzen der mobilen genetischen Elemente in ihrer Gesamtheit noch als Junk-DNA bezeichnet. Angesichts der heutigen Erkenntnisse zur Biologie von mobilen genetischen Elementen sowohl hinsichtlich ihrer zunächst eigennützigen Aktivität innerhalb von Genomen als auch ihrer Relevanz für die Biologie von Genomen, und damit von Organismen, ist diese Bezeichnung keinesfalls mehr gerechtfertigt. Die Zukunft wird sicherlich noch aufregende und auch für die Biologie des Menschen wichtige Befunde zu mobilen genetischen Elementen liefern.
Literatur Bild: © istockphoto.com| gremlin |
L&M 3 / 2015
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Der Autor:NewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |